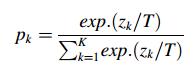利用高光谱技术对食品领域白色塑料的鉴别
背景
塑料根据其可降解性分为可降解塑料和不可降解塑料。不可降解塑料制品由于价格低廉,在日常生活中被广泛使用。然而,大多数不可降解的塑料制品不能及时有效地处理。造成环境严重污染,造成生态破坏、作物减产、食品**问题。目前,可降解塑料的推广应用才刚刚起步,许多不合格的塑料制品混入市场。这给市场监管带来了很大的困难,因此迅速识别不可降解和可降解塑料势在必行。
高光谱成像技术因其无损、无污染、低耗、**等优点,在质量控制、定量分析、定性鉴定等方面得到了广泛应用。普通塑料聚合物具有不同的光谱特征,因此利用可见近红外波段对塑料聚合物进行识别是可行的。塑料光谱信息采集技术的进步也推动了光谱数据挖掘方法的**。常用主成分分析、小波变换提取敏感特征,再用偏*小二乘判别分析、支持向量机、随机森林等方法进行分类。然而,传统的机器学习方法通常需要人工进行特征提取和分类器设计,难以灵活地处理分类任务。
与传统机器学习相比,深度学习具有端到端特征,避免了复杂的特征工程。代表性的模型是卷积神经网络(Convolutional neural networks, CNN)和残差网络(Residual networks, ResNet)。利用ResNet中提出的残差连接直接连接不同的网络层,在保证良好网络性能的同时可以加深网络。然而,当CNN或ResNet的深度和宽度固定时,由于卷积核不容易根据实际数据的特点进行有效调整,其表示能力将受到限制。动态卷积是在静态卷积的基础上改进的非线性运算。它可以解决深度学习模型适应性差的问题,更有效地提取数据特征。同时,动态卷积计算是高效的,因为并行卷积核通过聚合共享输出通道,这不会增加网络的深度和宽度。在网络中引入动态卷积可以在特征识别过程中自适应捕获特征。
为此,我们提出了一种塑料高光谱数据挖掘方法,主要贡献如下:
(1)设计了白色可降解和不可降解塑料的识别策略。
(2)提出了一种基于动态残差网络(Dy-ResNet)的高光谱数据挖掘模型。在模型优化过程中引入残差模块,避免梯度消失;引入动态卷积层,提高模型的自适应性和表示能力。
(3)Dy-ResNet有效区分了白色可降解塑料和不可降解塑料。为食品领域中可降解和不可降解塑料的识别提供思路。
试验设计
东北电力大学门洪教授团队利用GaiaSorter高光谱成像系统(江苏双利合谱公司)获取了17类可降解塑料和不可降解塑料共计1020个样本的高光谱影像(图1)。其波长范围为380 ~ 1038 nm,波段数为520。在每个样本的高光谱图像的中心区域选择一个像素大小为60 × 60的采样区域作为感兴趣区域,获取其平均光谱,并对其进行SG滤波和SNV预处理。
CNN主要由卷积层、池化层和全连接层组成,其中全连接层的核心操作是卷积和池化。卷积提取了多波段高光谱特征,同时充分保持了特征之间的位置关系。池化可以减小特征图的大小,可以保留*关键的信息,有效防止网络过拟合。同时,通过池化可以减少网络中的节点数量,有效提高了网络的计算效率。ResNet由两个卷积层组成,卷积核大小为3 × 3,填充和步长为1(图2)。将原始特征与挖掘特征相结合,可以有效避免塑料高光谱图像深度特征提取中的特征退化问题。
本文将静态卷积层替换为动态卷积层,在不增加网络深度和宽度的前提下提高了计算效率。根据卷积核的自适应性,动态卷积层具有鲁棒的表示能力。动态卷积层结构如图3所示。首先,对全局位置信息进行平均池化压缩;然后,将压缩后的信息通过全连接层映射到激活层,其中ReLU为激活函数。激活信息通过全连接层映射到softmax层。*后,softmax层输出K个权值用于核聚合。K表示参与核聚合的卷积核的个数,K的增加会导致模型的复杂度增加。在大多数情况下,softmax层的输出值相对稀疏,因此只有一小部分卷积核可以跨层优化,导致神经网络在训练过程中收敛缓慢。因此,减少softmax层中的注意力是必要的,这样可以使训练更有效。计算公式如1所示,当T = 1时,公式为原始softmax层。利用Dy-kernel自适应提取高光谱数据的特征,然后依次通过BN和ReLU得到*终输出。
|
|
(1) |
本文提出了自适应提取塑料高光谱数据特征的Dy-ResNet方法,该方法主要由包含动态卷积层的残差块组成。图4显示了Dy-ResNet的结构,包括一个卷积层、一个BN层、四个动态卷积残差块(DR-block)、两个池化层、一个flatten层和一个全连接层。
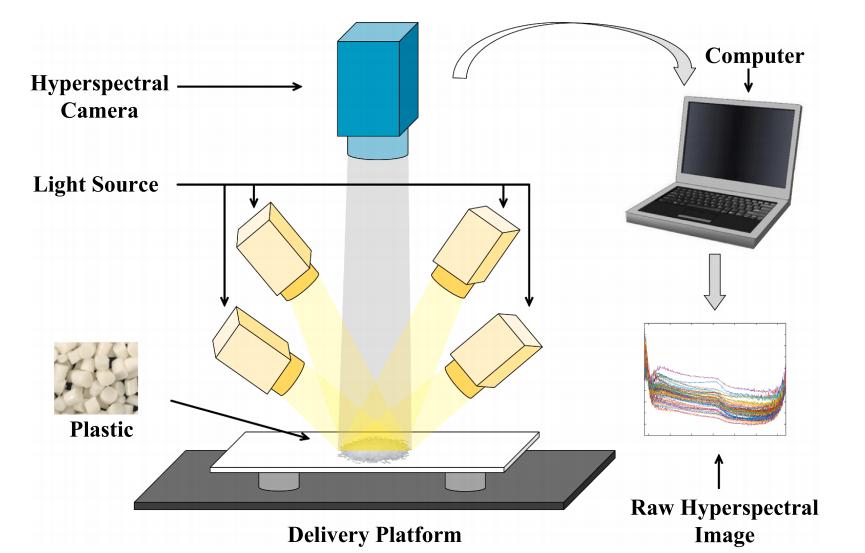
图1 高光谱塑料检测系统
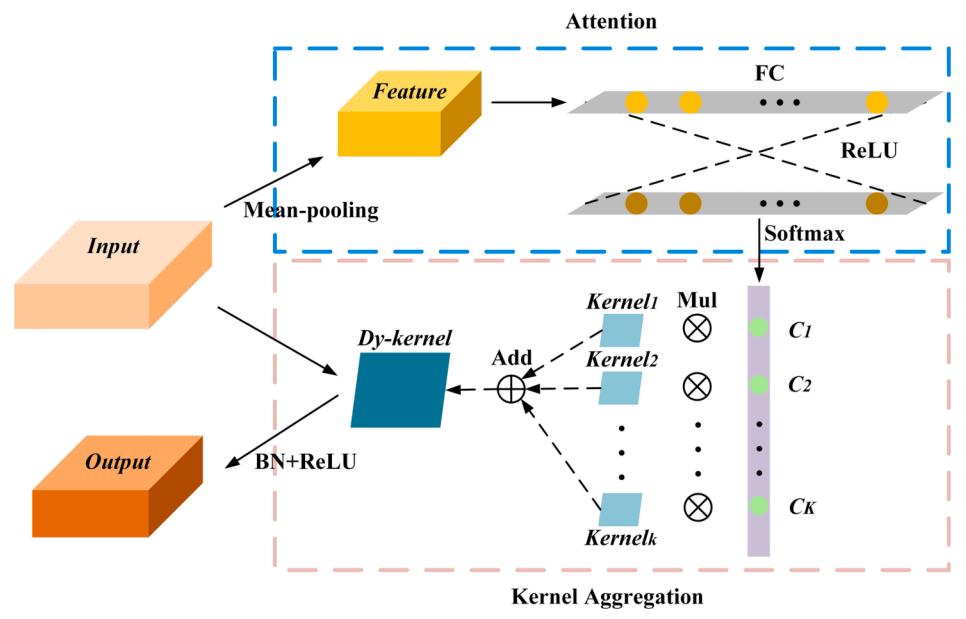
图2 残差块结构
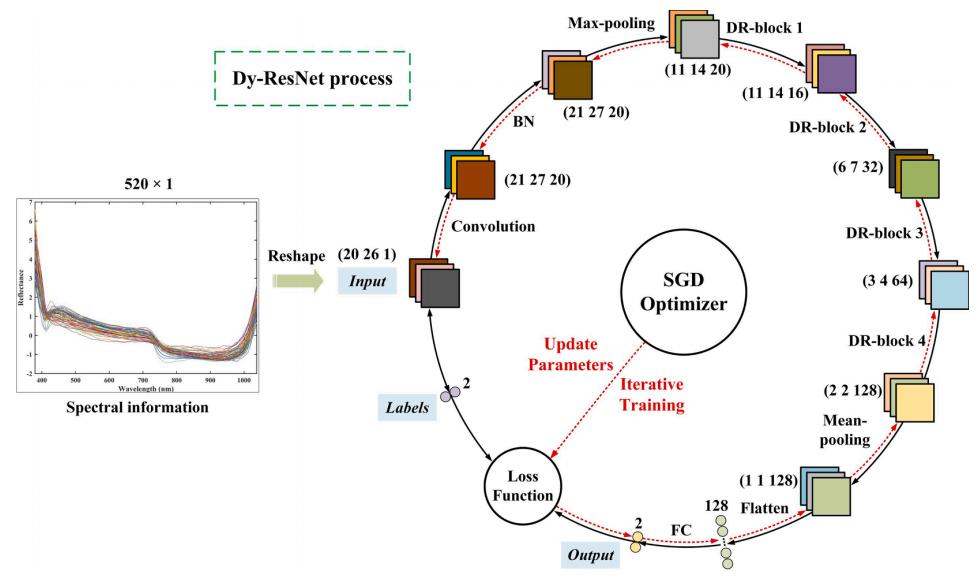
图3 动态卷积层结构
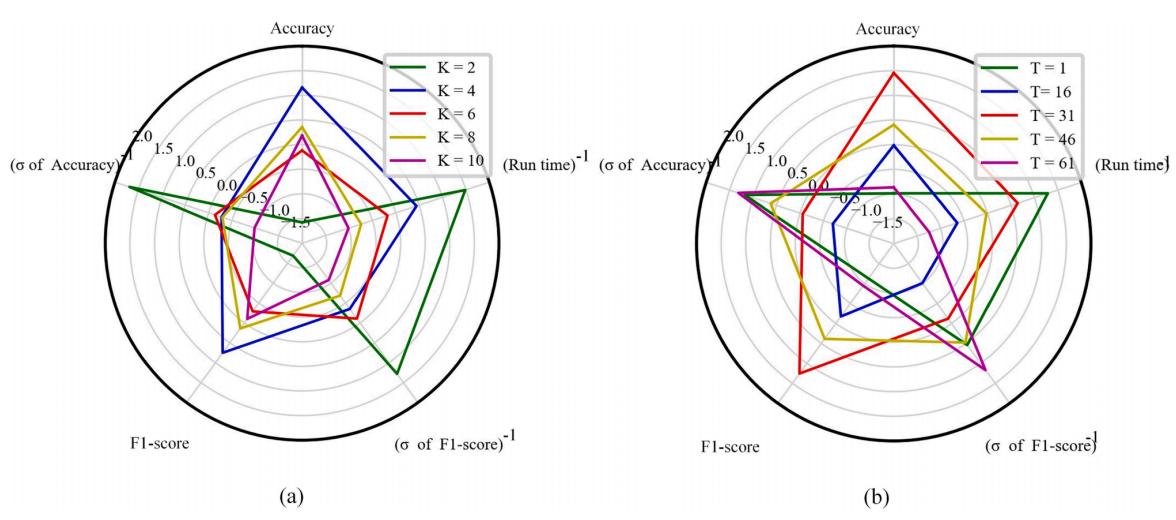
图4 Dy-ResNet的结构
结论
在动态卷积层中,参数K和T显著影响稳定性和精度。如果K过大,虽然模型具有更强的表达能力,但优化所有卷积核和注意力更加困难,网络更容易出现过拟合。在softmax层中,T控制了注意力权值的稀疏性,适当的T可以使模型在早期训练中更有效。因此,有必要确定K和T的*优值。如图5所示,当K较小时,模型的训练时间和分类稳定性都较好。然而,K的数量限制了动态卷积核的自适应能力,降低了模型的分类精度和F1-score。当K设置为4时,既保证了模型的分类性能,又充分考虑了训练时间。T越大,模型的稳定性越高。但是,当T过于大时,模型的分类性能会下降,因此将T设为31。

图5 不同参数的动态卷积层
可降解和不可降解塑料的分类结果如表1所示。虽然Dy-ResNet、ResNet、Dy-CNN和CNN模型都取得了很好的分类效果,但提出的Dy-ResNet模型的识别效果更好。Dy-ResNet模型的识别准确率为99.06%,F1-score为98.86%,Kappa为97.73%。此外,对比CNN和ResNet模型的分类结果可以看出,残差连接的引入更有助于挖掘高光谱数据的深层特征。Dy-ResNet模型的精度、F1-score和Kappa分别比ResNet模型高1.53%、1.85%和3.71%,表明动态卷积层可以有效提高模型在高光谱数据挖掘中的适应性和表征能力。
不同模型的混淆矩阵如图6所示。可以看出,四种分类模型的分类效果都很好,说明该分类模型是有效的。此外,可以看到Resnet、Dy-CNN和CNN模型将一些不可降解塑料预测为可降解塑料,这可能是由于训练集中样本数量不平衡造成的。然而,Dy-Resnet模型可以避免由于训练集各类别样本数量不平衡而导致的错误预测。
表1 四种网络模型的2-分类实验结果
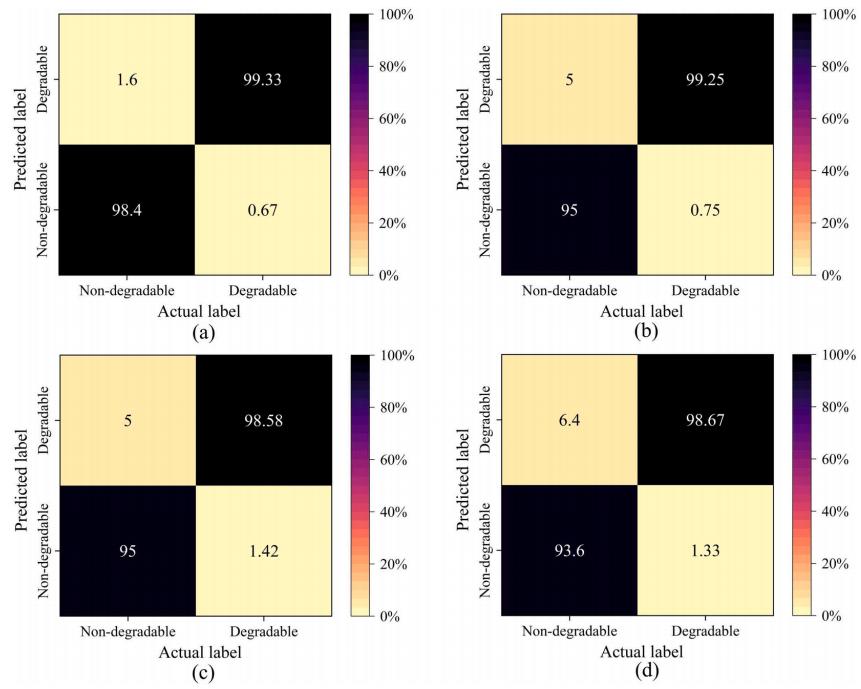
图6 四种方法对可降解塑料和不可降解塑料的分类结果。Dy-ResNet(a), Dy-CNN(b), ResNet(c), CNN(d)
为了进一步验证Dy-ResNet在塑料识别中的有效性,分别使用Dy-ResNet、ResNet、Dy-CNN和CNN模型对17种样品进行分类。4个模型在全连接层的输出神经元数量从2个变为17个,4个模型的其他结构和超参数不变。17种塑料的分类结果见表2。总体而言,DyResNet的分类性能优于其他模型,其准确率为89.76%,F1-score为89.68%,Kappa为89.13%。与CNN相比,Dy-ResNet中的残差连接可以有效提取塑料高光谱图像的深层特征。在5个独立实验中,Dy-ResNet的模型精度*高,表明其具有*好的识别稳定性。残差连接和动态卷积层的引入可以有效地提高模型的稳定性。
测试集样本的预测标签与实际标签形成的混淆矩阵如图7所示。可以看出,这四种模型都能有效识别标记为0、6、7、8、9、11、12、13、14、15的样本。但值得注意的是,四种模型在识别标签2和标签10的样品时存在一些错误,主要是将标签2的样品部分归类为标签3,将标签10的样品部分归类为标签11。这主要是由于,除了碳酸钙外,标签2和标签3对应的样品在成分材料上是相同的。除淀粉外,标签10和11对应的样品在组成材料上是相同的。上述样品中相似的成分使得所收集的高光谱图像难以区分,这就是它们被错误分类的原因。
表2 四种网络模型的17种分类实验结果

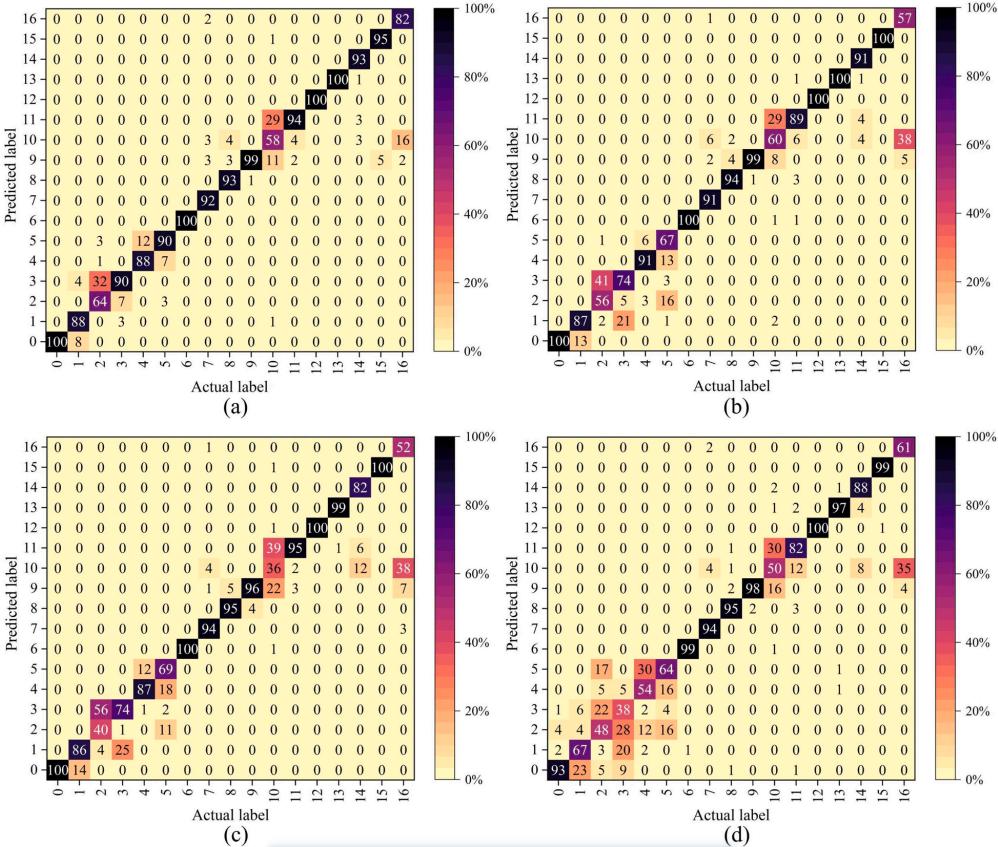
图7 四种方法对17种塑料的分类结果。Dy-ResNet(a), Dy-CNN(b), ResNet(c), CNN(d)
合理解释本研究提出的方法在塑料识别任务中取得良好的效果十分必要。Grad-CAM可用于可视化基于CNN模型的模型识别过程。如图8(a)所示,在区分可降解和不可降解塑料时,Dy-ResNet提取的特征光谱波段分布在可见光和近红外范围内。此外,用于识别不可降解塑料的关键特征主要分布在580 ~ 860 nm范围内,用于识别可降解塑料的关键特征主要分布在680 ~ 1030 nm范围内。如图8(b)所示,Dy-ResNet提取的特征光谱波段在区分17种塑料时也分布在可见光和近红外范围内。总体而言,用于识别塑料种类的特征光谱波段主要集中在560 ~ 860nm范围内。然而,每种塑料的特征光谱波段的分布范围是不同的。例如,标记为15的样品的特征光谱波段主要集中在可见光范围,而标记为12的样品的特征光谱波段主要集中在近红外范围。因此,选择380 ~ 1038 nm光谱波段进行塑料识别,有助于充分探索不同塑料在不同光谱波段的特性。

图8 用于塑料识别的特征光谱可视化。2分类实验(a), 17分类实验(b)
作者信息
门洪,博士,东北电力大学自动化工程学院教授,博士生导师。
主要研究方向:智能感知与模式识别。
参考文献:
Xia, X., Wang, M., Shi, Y., Huang, Z., Liu, J., Men, H., & Fang, H. (2023). Identification of white degradable and non-degradable plastics in food field: A dynamic residual network coupled with hyperspectral technology. Spectrochim Acta A Mol Biomol Spectrosc, 296, 122686.
https://doi.org/10.1016/j.saa.2023.122686